6.2.1
ETAPAS DE SÍNTESIS DEL ARN
- Etapas de la transcripción
Al igual que en procariotas, las
etapas básicas son la
iniciación, la Elongación
y la terminación. Los procesos enzimológicos también son los
mismos, por lo que vamos a ver cuáles son las diferencias esenciales que
caracterizan al proceso eucariótico.
Hasta no hace mucho, los promotores se
analizaban por deleciones seriadas desde el extremo 5' del gen, por lo que se
consideraba que el promotor empezaba a partir del nucleótido más distal cuya
deleción provocaba la pérdida de la capacidad transcriptora. Siguiendo este
criterio, quedaban excluidos del promotor muchos elementos reguladores que no
influían decisivamente sobre la transcripción del gen.
Para la transcripción se necesitan una
serie de factores de transcripción que se nombran empezando por la abreviatura
TF, le sigue el número de la polimerasa que reconoce, y termina por una letra
que sirve para distinguirlos unos de otros. Por razones históricas, existen
muchas excepciones a esta regla.
Iniciación
Durante la iniciación es cuando se
produce el reconocimiento del promotor y asociación de la polimerasa. Es la
parte más compleja de la transcripción y sobre la que se ejercerán la mayoría
de las actividades reguladoras. Debido al gran tamaño del genoma, los factores
de transcripción y la polimerasa tienen muy difícil reconocer sus sitios
específicos. Para conseguir especificidad se requieren mecanismos
másintrincados como el súper enrollamiento—condensar las regiones de DNA que no
se tienen que expresar
—con el objeto de disminuir la cantidad de DNA a rastrear. Como
se verá, la presencia de los nucleosomas entorpece la transcripción de los
genes.
La función del promotor esencialmente
consiste en facilitar la unión de la RNA-polimerasa al DNA y asegurar que el
inicio de la transcripción se realice con mucha eficacia y en un nucleótido
determinado. El complejo de iniciación produce en la región de inicio de
transcripción el desenrollamiento y separación de las dos cadenas del DNA (complejo abierto) que es donde va a tener lugar la síntesis de la cadena de RNA. Esta
estructura se desplazará por el DNA a medida que avanza la elongación (burbuja detranscripción). Es necesario que se en torno a
la RNA-polimerasa forme el complejo holoenzimático
polimerasa para que se pueda empezar a transcribir. El
modelo más aceptado propone que en la región promotora se van asociando los
factores de transcripción y subunidades de la Holoenzima de manera ordenada y
constante, hasta que se une la polimerasa central. También se requiere la
presencia de una DNA-helicasa y la estabilización
de la región desapareada con proteínas específicas. El proceso enzimático por
el que se añaden los ribonucleótidos uno a uno a partir del primer nucleótido
trifosfato es igual que el procariota.
1.- Decondensación
del DNA para la transcripción
Para poder iniciar una transcripción,
la maquinaria transcriptora ha de poder acceder a la hebra de DNA, lo que
implica la descondensación previa de la región implicada del cromosoma. Este
fenómeno se reconoció por primera vez al estudiar citoquímicamente los
cromosomas politécnicos de las glándulas salivares de Drosophila.
En los cromosomas politécnicos se distinguen
una serie de bandas muy características y constantes. Cada banda contiene unos
pocos genes (menos de 10) y desaparece cuando uno de estos genes se transcribe
activamente, ya que se desempaqueta el DNA.
Para que la
maquinaria transcripcional pueda acceder al promotor, éste ha de encontrarse
descondensado (como máximo, debe estar en forma de fibra de 10 nm) y
desprovisto de nucleosomas (se detectan como regiones hipersensibles a las
nucleasas). En cambio, en esta región abundan las proteínas no histónicas como
HMG14 y HMG17. La eliminación de los nucleosomas se debe al ensamblaje y
desensamblaje de los mismos controlado por la acetilación de las histonas.
La acetilación de los aminoácidos Lys y Arg (que aportan las cargas positivas)
produce grupos amida que no se protonan a pH fisiológico, por lo que estas
proteínas pierden su carga; tradicionalmente se pensaba que con ello se
disminuye su afinidad por el DNA, pero parece más probable que la acetilación
no influya en la interacción con el DNA sino entre las propias histonas del
nucleosoma. Las actividades histona-acetil-transferasa e histona-desacetilasa
se encargan de realizar este proceso. Cada vez es más evidente que los factores
de transcripción tienen actividad acetil-transferasa para facilitar la
liberación de los nucleosomas en torno a la secuencia que reconocen.
Durante la
elongación de la transcripción, la RNA-polimerasa es capaz de desplazar los
nucleosomas en la zona codificante: cuando la RNA-polimerasa se encuentra con
uno, lo empuja sin esfuerzo los primeros 30 nt y luego se ralentiza hasta el nt
80 —con paradas cada 10 nt—, momento en el que el nucleosoma es desplazado por
detrás de la enzima gracias a la energía potencial topológica que se acumula en
las supe hélices por la tensión del DNA.
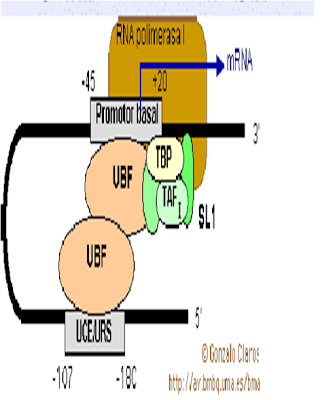
Inicio
de la RNA-polimerasa I
Esta polimerasa, a diferencia de las
otras dos, va a sintetizar un único tipo de transcrito, el pre-rRNA grande de
13,7 kb en los mamíferos (45S). El promotor de cada unidad detranscripción (que
está repetida miles de veces en la mayoría de los genomas) abarca unas200 pb
delante del inicio de transcripción y comprende dos regiones:
1.- El promotor basal, que va de la posición – 45
hasta la +20, por lo que incluye el inicio de transcripción (+1). Esta región
es rica en G y C(salvo la parte del Inr alrededor de la posición +1, que es
rica en A y T) y se necesita para iniciar correctamente la transcripción,
siendo capaz de promover la transcripción por sí misma. No existe ninguna secuencia
conservada entre especies — ya que
sólo hay un promotor de estos porespecie — que pueda servir de señal de reconocimiento.
2.- La secuencia reguladora distal
(UCE: upstream control element o URS, upstreamregulatory
sequence) se sitúa en la parte 5’ del gen, entre las
posiciones – 107 y – 180. Sueliminación hace que la transcripción descienda hasta 100 veces.
Su secuencia es un 85%homóloga a la del promotor basal y también rica en GC.
La
falta de homologías en las secuencias promotoras de este tipo de genes hace que
sean promotor es muy específicos de especie y no sean reconocidos por la
maquinaria de transcripción de especies heterólogas.
Los factores que reconocen este
promotor para facilitar la entrada de la RNA-polimerasa I son:
1.- UBF ( upstream binding factor ): es un homodímero que reconoce la UCE y provoca un giro de 360° que
acerca esta región al promotor basal, con lo que mejora la eficacia de la
transcripción al interaccionar con el otro factor de transcripción (SL1). Otros
autores proponen que UBF reconoce a la vez el promotor basal y el UCE, y por
eso induce el giro en el DNA.
2.- SL1 (selective factor, también denominado
TIF-IB o Rib1 en otras especies): es unheterotetrámero formado por la TBP (TATA
binding protein) y tres TAF I (TBP
associatedfactors). La unión del SL1 reconoce el promotor como funcional y
permite que se una la RNA-polimerasa I para comenzar la transcripción; por eso,
la función del SL1 recuerda la del factor σ de la RNA polimerasa bacteriana.
La TBP se aisló inicialmente de los promotores de
la RNA-polimerasa II como la proteína que reconocía específicamente la caja
TATA en estos promotores.
Los TAF I
sí suelen ser específicos de los promotores. El subíndice indica
el tipo de promotor al que se unen. Las distintas moléculas de TAF del mismo
factor de transcripción suelen ser distintas. Algunos TAF se ha visto que
también forman parte de otros complejos proteicos que están directa o
indirectamente relacionados con la regulación de la transcripción—como
lashistona-acetil-transferasas— . Los TAF se asocian entre
sí mediante el dominio HFD ( histone fold domain ) de la misma forma que
se asocian las histonas.
Por
tanto, se deduce que el SL1 se une al promotor de forma similar a como lo hacen
el DNA y los nucleosomas.
Inicio de la RNA-polimerasa
III
Ninguno de los genes que transcribe
esta polimerasa produce una proteína, sino RNA funcionales, y son pequeños
(menores de 300 nt): pre-rRNA 5S; los tRNA; algunos RNA pequeños que
intervienen en el ayuste—snRNA U6, 7SK, 7SL, 4,5S y M—y los microRNA. Todos
comienzan con una G y se localizan sobre la zona codificante (no delante como
todos los demás) en lo que se denomina la región interna de control ( ICR : interna
al control región ). Cadena arriba hay una secuencia
necesaria para la especificidad y eficacia del sitio de inicio: la secuencia reguladora proximal (URS : upstream regulatory sequence ).
Inicio de la RNA-polimerasa
III
Ninguno de los genes que transcribe
esta polimerasa produce una proteína, sino RNA funcionales, y son pequeños
(menores de 300 nt): pre-rRNA 5S; los tRNA; algunos RNA pequeños que
intervienen en el ayuste—snRNA U6, 7SK, 7SL, 4,5S y M—y los micro RNA. Todos comienzan
con una G y se localizan sobre la zona codificante (no delante como todos los
demás) en lo que se denomina la
región interna de control ( ICR : internal control región
). Cadena arriba hay una secuencia necesaria para la
especificidad y eficacia del sitio de inicio: la secuencia
reguladora proximal ( URS : upstream regulatory sequence ).
Según se coloquen la ICR y la URS, se
distinguen tres tipos de promotores:
De
tipo «a» (como el de la figura) El del rRNA 5S. La ICR incluye dos cajas (
A y C ), de unos 20 pb cada una, entre las regiones +55 y +80, que son
reconocidas inicialmente por el factor TFIIIA. La presencia del TFIIIA provoca
una curvatura en el DNA y a continuación atrae al factor TFIIIC. Con el consumo
de ATP, se une el factor TFIIIB que también necesita la existencia de la caja
A, pero no de la C. Con la entrada del TFIIIB en presencia de TFIIIA y TFIIIC
se ha formado el complejo de preiniciación con lo que puede incorporarse la polimerasa. El complejo de
preiniciación indica que el gen está listo para ser transcrito, es un complejo
estable y puede mantenerse a lo largo de varios ciclos de replicación; in vitro, el promotor funciona con tal de que el
TFIIIB ya estuviera unido.
De
tipo «b»
El de los tRNA, snRNA y posiblemente los microRNA. No tienen caja C, pero
tienen una caja B en posiciones variables y una caja A más cercana al inicio de transcripción, y no
necesitan el TFIIIA. La interacción del TFIIIB sólo depende de la presencia de
la caja A.
De
tipo «c» El
del gen del snRNA U6 y 7SK. No son intragénicos—no presentan IRC— sino que se encuentran
en la región 5' del gen, contienen una caja TATA a –
25, que es lo que reconoce el TFIIIB, y la URS está en –60. Necesitan que el factor detranscripción SNAPc se una al TFIIIB
para que luego se una la polimerasa. Durante muchos años se dudó de que fuera
la pol-III y no la pol-II la que los transcribía.
El TFIIIA es una proteína de 37 kDa con 9 dedos de zinc como
dominio de unión al DNA en su región N- terminal, que
ocupa casi toda la proteína.
El TFIIIC es un complejo proteínico de más de 500
k Da con al menos 5 subunidades diferentes.
El TFIIIB es equivalente al SL1, pues se trata de
un heterotetrámero formado por la TBP y tres TAF III
diferentes entre sí y a los vistos para el SL1. TFIIIB sitúa la
RNA-polimerasa III correctamente en el inicio de transcripción sobre la URS.
Los promotores de la RNA-polimerasa
II
Los genes transcritos por la
RNA-polimerasa II son todos los mRNA y muchos snRNA implicados en su ayuste,
por lo que son los responsables de las proteínas de la célula. Los promotores
se localizan siempre en la zona 5' no codificante previa al sitio del inicio de
la transcripción.
Aunque la estructura de estos
promotores es bastante dispar, se pueden definir los tres elementos esenciales
de este tipo de promotores:
1.- Promotor basal o mínimo: es el que determina el sitio de inicio de transcripción. En
esta parte se encuentran varios elementos comunes a todos los promotores. Éstos
son la Caja TATA
para verificar que es un promotor, la secuencia iniciadora
Inr para determinar el inicio de transcripción, y el amentó de
respuesta al TFIIB (BRE ) para orientar el promotor.
2.- Los elementos proximales: se encarga de
determinar la frecuencia con la que se debe iniciar la transcripción. Los
elementos proximales más importantes son la caja CAAT, las
cajas GC y las secuencias distales específicas ( SDE
).
3.- Los elementos distales: se encuentran a más
de 1 kpb del inicio de transcripción, tanto hacia 5' como hacia 3', y funcionan
independientemente de su orientación. Los hay potenciadores y silenciadores.
Muchos autores prefieren definir los
promotores sólo como los elementos basales y proximales, dejando a los terceros
exclusivamente como secuencias reguladoras.
Inicio de los promotores de la RNA-pol
II
La RNA-polimerasa II interacciona
simultáneamente con gran variedad de factores de transcripción. Según se unan a
cada uno de los elementos descritos anteriormente, se puede hablar de factores
detranscripción generales (reconocen los elementos basales),
Proximales (reconocen los elementos proximales)
e inducibles (reconocen los elementos
distales y los SDE). La forma y el orden en que se unen determina el inicio de
la transcripción.
Factores
de transcripción generales y cómo se unen
Los factores generales, al reconocer
las secuencias más conservadas, son a su vez, los más conservados. Promueven la
formación del complejo de inicio, mediando la fijación de la polimerasa y el correcto
punto de comienzo de transcripción. Véase un esquema
general del proceso.
La secuencia de unión de estos
factores comienza con la unión del
TFIID al surco menor del DNA (libre de
histonas) a través de la caja TATA (o del MTE/DPA) a través de la TBP, protegiendo
unos 35 pb. Esta etapa es la crítica en
el proceso. El TFIID está muy conservado y parece ser el único que reconoce específicamente
una secuencia de DNA, la caja TATA, aunque también se une a promotores que no
la tienen, uniéndose, en tal caso, al elemento Inr. Su tamaño es de unos 800
kDa. Uno de sus componentes es la TBP que aquí sí reconoce específicamente la
caja TATA. La TBP tiene un dominio de
unión a DNA un poco especial que se conoce como «silla de montar». In vitro
basta la TBP para iniciar una transcripción. Los otros componentes del TFIID
son varias moléculas de TAF II
(de 9 a 12 distintas) que varían enormemente de unos promotores a
otros. Una buena parte de las TAF son proteínas reguladoras y factores
específicos de tejido u órgano y otras median entre el complejo basal de la
RNA-polimerasa y algunos reguladores. Por tanto, las TAF II
son necesarias para responder a la regulación de los promotores.
Factores
de transcripción proximales
Son los que contribuyen a aumentar la eficacia de la
formación del complejo de inicio, o bien favorecen la
actividad de la propia polimerasa. Cada promotor necesita un conjunto preciso
de estos factores para su funcionamiento óptimo. A modo de ejemplos:
1.- CTF (también conocido como NF1 ):
interacciona con la caja CAAT y hace aumentar la eficacia del promotor. El
dominio que hace de activador es del tipo rico en Pro (20%) de la secuencia.
2.- CBP, ACF, CP1
y CP2 también interaccionan con la caja CAAT
en distintos tipos celulares, por lo que se sugiere que debe existir una
familia de de proteínas capaces de discriminar las distintas cajas CAAT.
3.- SP1 (monómero de 105 kDa): se une a las cajas GC, pero no a todas, por lo
que las secuencias adyacentes han de influir de alguna manera en esta interacción,
ya que su unión protege unos 20 nt, cuando el motivo GC es sólo de 6-8 nt.
Contacta con TFIID, activando la transcripción. El motivo de unión a DNA de SP1
son 3 dedos de Zn en la zona C-terminal. El dominio activador tiene un 25% de
Gln.
Factores
de transcripción inducibles o distales
Los factores que reconocen los
elementos distales
facilitan (o impiden) la formación del complejo de inicio de transcripción. A diferencia de los anteriores, sólo están presentes
en momentos específicos o en células que lo requieren. O sea, son reguladores y se verán ejemplos característicos en
el tema de la regulación eucariótica.
Lo habitual es que el contacto no sea
directo entre el factor distal y la polimerasa, sino que esté mediado por un coactivador que interacciona simultáneamente con la polimerasa y con el factor
distal. Uncoactivador se define como «aquellos factores que se necesitan para
ejercer el efecto de los activadores que se unen al DNA, pero que no son
necesarios para la transcripción basal per se, ni muestran una unión específica
de sitio por sí mismos».
Los coactivadores suelen estar
formados por muchas subunidades proteicas. Puesto que el mismo coactivador
puede recibir señales de distintos potenciadores, son distintas subunidades de
éste las que van a interaccionar con los elementos distales. Uno de los
coactivadores holoproteicos más conocidos en humanos recibe el nombre de Mediador. Se trata de
un complejo de más de 20 subunidades (más grande que la propia RNA-polimerasa)
tan conservado que es capaz de reconocer señales que provienen de activadores
de distintas especies. Se ha visto que se organiza en módulos que se pueden
disociar del conjunto (coincidiendo con los colores de los dibujos anteriores y
con la recogida y envío de las señales).
En la figura de la derecha se
esquematiza la estructura del complejo mediador y a la derecha cómo interacciona
con la RNA-polimerasa y con el DNA.
Elongación
La Holoenzima ha permanecido inmóvil
mientras se polimerizan los primeros nucleótidos, hasta que el TFIIK fosforila
el CTD, activado por el TFIIE. Se ha visto que este dominio fosforilado se
ubica muy próximo al canal por el que sale el RNA que se está sintetizando, por
lo que se piensa que interviene en la transferencia de factores proteicos al
mRNA naciente. Tras la fosforilación, la polimerasa se libera de todos los
factores de iniciación excepto la helicasa del TFIIF y comienza su avance por
el DNA. En torno al elemento Inr pueden quedarse los TFII A, B y D así como
otros activadores de la transcripción, listos para reiniciar otro ciclo de
transcripción—esta vez más abreviado puesto que las primeras estapas no han de
repetirse—.
Una actividad helicasa sigue abriendo
el DNA por delante, lo que genera importantes tensiones en la molécula que se
compensan por la acción de las topoisomerasas. Por detrás de la polimerasa el
RNA recién sintetizado se va desapareando del DNA molde y quedando en forma de
cadena sencilla de RNA. Las dos cadenas de DNA se vuelven a aparear y gracias a
la asistencia de otra topoisomerasas, serebobina de nuevo el DNA. En esta etapa
intervienen al menos
16 factores de elongación cuyo mecanismo
exacto se desconoce. Su función siempre aparece relacionada con 3 fenómenos
principales:
1.- La eliminación de las pausas o
paradas de la polimerasa. Los mejor identificados son TFIIS (antes llamado SII) que sirve para
evitar que la RNA-polimerasa se detenga prematuramente (limita las pausa de la
RNA polimerasa. La presencia de este factor también se necesita para que se
estimule la actividad editora.) y por los factores que alteran la Km y la Vmax
de la enzima para incrementar su capacidad catalítica: ELL, CSB
y SIII
(elonguina).
2.- Para que la polimerasa funcione,
tiene que estar fosforilado
hSPT5. Gracias a este factor se recluten las enzimas
necesarias para la adición de la caperuza: también sirve para que se incorpore
TAT-SF1 que es necesario para que se reclute en los sitios adecuados la
maquinaria del ayuste. También se incorporan las proteínas que intervienen en la
remodelación de los nucleosomas durante la elongación (SWI/SNF, FACT, HMG14,
elongator...), ya que sigue siendo necesario despejar el camino.
3.- El mantenimiento del estado
fosforilado del CTD (
PTEFb y otros), ya que sudesfosforilación inhibe la elongación. Este factor
también mantiene fosforilado el hSPT5.También sirve para reclutar otros
factores como AT-SF1.
Está claro que la elongación, la
terminación y la maduración del mRNA están íntimamente interconectados y
necesitan estar coordinados con precisión.
Terminación
Las secuencias terminadoras
propiamente dichas están aún poco caracterizadas, pero se ha visto que provocan
dos efectos:
1.- detención o pausa en el avance de
la polimerasa al pasar por ellas.
2.- desestabilización del híbrido
RNA—DNA.
El resultado final es la separación de
los componentes del complejo de transcripción (incluida ladesfosforilación del
CTD de la RNA-polimerasa II) y desensamblaje de la Holoenzima para ser
reciclada en otro complejo de iniciación. El extremo 3’ de los RNA eucarióticos
transcritos no coincide con el extremo 3’ de los RNA maduros ya que es habitual
que los extremos de los transcritos primarios se corten durante la maduración
(excepto los de la RNA-polimerasa III). También se comprende la dificultad del estudio
de las secuencias y mecanismos de terminación, ya que los transcritos primarios
(hnRNA y otros pre-RNA) son de corta vida y difícil purificación.
Terminación
de la RNA-polimerasa I
Los rRNA se expresan mucho debido al
confinamiento de estos genes en el nucléolo, la ausencia de nucleosomas en su
región codificante y el estar repetidos en tándem. Por otra parte, el
acoplamiento entre el fin de la transcripción de un pre-rRNA con el comienzo
del siguiente (posición en tándem) acelera aún más esta transcripción: cuando
la terminación de la transcripción de un gen acaba cerca del promotor del
siguiente, se activa la transcripción del segundo gen ya que resulta más
rentable polimerizar secuencia inútil que soltar el DNA y volver a iniciar otra
transcripción. La señal de terminación de los rRNA es una secuencia de 18 pb
duplicada que se encuentra triplicada, de la que hay dos copias cerca del extremo 3’
del gen 28S (T1 y T2) y la tercera a 200 pb del inicio de transcripción
del gen siguiente (T3).
En una secuencia indeterminada se une
una proteína terminadora (
Reb1p en las levaduras o TTF-I en los mamíferos) que detiene el avance
de la RNA-polimerasa. Una vez detenida, una región rica en T en la cadena
codificante facilita la separación del RNA. No se ha observado que se formen
horquillas.
Terminación
de la RNA-polimerasa II
Esta enzima tiene un mecanismo
equivalente al de la terminación dependiente de ρ de procariotas. Se ha observado que la enzima
se detiene en la región de terminación debido bien a secuencias específicas en el
DNA, o bien a proteínas que favorecen la pausa que aún no se han identificado.
Una proteína similar a ρ se une al RNA e induce la
separación del complejo holoenzimático. De hecho se ha comprobado que la propia
ρ de E. coli es capaz de
inducir esta terminación. Está directamente relacionada con la poliadenilación
del mRNA.
Terminación
de la RNA-polimerasa III
Esta enzima tiene un mecanismo
equivalente al de la terminación intrínseca en procariotas. La detención de la
RNA-polimerasa se produce en alguna de las T que hay al final del gen. Se
producen diferencias en la terminación dependiendo del tipo de promotor que se
ha utilizado en la iniciación:
1.- Si es un promotor de tipo «a», el TFIIIA atrae los factores de terminación
específicos que van a reconocer las T si van precedidas de un palíndromo de GC.
2.- Si es un promotor de
tipo «b» es TFIIIC
el que atrae los factores de terminación.
3.- Si es un promotor de tipo
«c», SNAPc es la que va a reclutar los
factores de terminación que van a reconocer las T dentro de una zona rica en A
puesto que se ha visto que la presencia de G inhibe la terminación en este
caso. A diferencia de la terminación de las otras dos
polimerasas, estos transcritos no van a sufrir ninguna modificación
en su extremo 3’, por lo que todos acaban en varias UUU.



















No hay comentarios:
Publicar un comentario